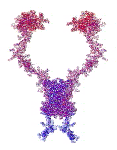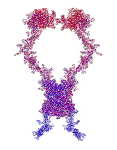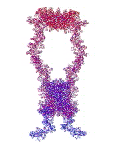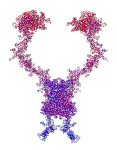|
Выпуск 633 Лаборатория Наномир Когда реальность открывает тайны,
уходят в тень и меркнут чудеса ... Начинается
изготовление модулятора на базе блокинг-генератора 



Skype: [10.11.2017 18:32:41] Мастер: Насчёт блокинг -а, может и будет, но Ваша схема не запустится. Впрочем это детали. Довести её можно и не сложно. Я сейчас не буду вдаваться, это мелочи.
Серьёзно другое, у Вас ведь были требования к импульсу, тут импульс будет очень "кривой", из за огромной индуктивности
[10.11.2017 18:32:44] Кушелев Александр Юрьевич: Дело тормозится как раз из-за нехватки денег у инвестора.
[10.11.2017 18:33:11] Кушелев Александр Юрьевич: А откуда индуктивность возьмётся?
[10.11.2017 18:34:13] Мастер: Индуктивность рассеивания трансформатора. Много витков, ибо сердечник придётся делать разомкнутым
[10.11.2017 18:34:30] Кушелев Александр Юрьевич: Есть конденсатор 0.5 мкф. Одну клемму нужно присоединить непосредственно к магнетрону. Вторую через модуляторную лампу ко второй клемме конденсатора.
[10.11.2017 18:34:35] Кушелев Александр Юрьевич: Какой
трансформатор?
[10.11.2017 18:34:58] Кушелев Александр Юрьевич: Модуляторная лампа открывает конденсатор на анод магнетрона.
[10.11.2017 18:35:18] Кушелев Александр Юрьевич: Трансформатор стоит в цепи обратной связи. Он нужен, чтобы открыть лампу, а потом закрыть.
[10.11.2017 18:35:41] Кушелев Александр Юрьевич: Какой
там будет фронт и спад не столь важно. Важно, чтобы полочка была ровной.
[10.11.2017 18:35:52] Кушелев Александр Юрьевич: А полочка определяется ёмкостю конденсатора
[10.11.2017 18:36:30] Мастер: Ну если фронт не важен, то да
[10.11.2017 18:36:46] Кушелев Александр Юрьевич: Хотя ... может быть должен быть отдельный
ключ, открывающий конденсатор для магнетрона и отдельный ключ, закрывающий.
[10.11.2017 18:37:19] Кушелев Александр Юрьевич: Фронт не важен. Важна только полочка, чтобы в течение микросекунды держался стабильный ток через магнетрон.
[10.11.2017 18:37:45] Кушелев Александр Юрьевич: Важно, чтобы фронт не убил магнетрон, т.е. чтобы он не был больше, скажем, пол микросекунды.
[10.11.2017 18:38:04] Кушелев Александр Юрьевич: Ну и спад тоже должен быть не больше. Иначе магнетрон сгорит.
[10.11.2017 18:38:20] Кушелев Александр Юрьевич: Короче, Вы сами это всё знаете.
[10.11.2017 18:38:28] Кушелев Александр Юрьевич: Меня интересуют сроки и цена
[10.11.2017 18:39:10] Кушелев Александр Юрьевич: Настроить Вы можете сами, если хотите.
[10.11.2017 18:40:57] Кушелев Александр Юрьевич: Правда, нужно ещё магнетрон достать. У Вас есть такая возможность? Его можно на время достать, скажем, на месяц. За это время мы сможем провести эксперименты и либо включить рубиновый генератор, либо не включить. В любом случае через месяц мы сможем либо вернуть магнетрон, либо купить.
[10.11.2017 18:45:24] Кушелев Александр Юрьевич: Я бы вообще сделал так:
1. Прислал бы Вам необходимые детали и аванс.
2. Прислал бы набор рубиновых шариков.
3. Как только Вы сможете засветить один шарик, тогда уже решать вопрос, как лучше действовать
дальше.
[10.11.2017 18:47:08] Мастер: Эээ, Александр Юрьевич, я работаю с техническим заданием, а не с шариком. Параметры импульса, за них я готов отвечать. Насчет магнетрона погонять - я подумаю, так сразу не решить. Ещё раз - мне ТЗ, от него плясать. Шарики засвечивать ... (продолжение
следует) Обсуждение
Антигомология = Программная аллотропия / Antihomology = Software allotropy Письмо, которое нужно разослать по лабораториям РСА Уважаемые коллеги! Для дополнительной проверки новой технологии определения структуры белка нам нужно определить структуры этих белков, точнее участков, состоящих преимущественно из поливалина n*Val: 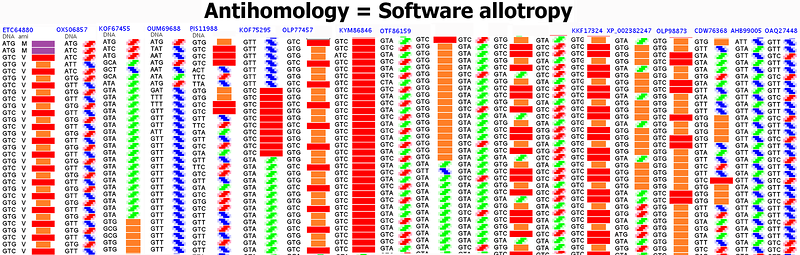 ETC64880: https://www.ncbi.nlm.nih.gov/nuccore/564568708
OXS06857: https://www.ncbi.nlm.nih.gov/nuccore/1226034760
KOF67455: https://www.ncbi.nlm.nih.gov/nuccore/918287724
OUM69688: https://www.ncbi.nlm.nih.gov/nuccore/1198313040
PIS11988: https://www.ncbi.nlm.nih.gov/nuccore/1277217132
KOF75295: https://www.ncbi.nlm.nih.gov/nuccore/918302080
OLP77457: https://www.ncbi.nlm.nih.gov/nuccore/1129165616
KYM86846: https://www.ncbi.nlm.nih.gov/nuccore/10 … mask=65536
OTF86159: https://www.ncbi.nlm.nih.gov/nuccore/CM
… temID=1362
KKF17324: https://www.ncbi.nlm.nih.gov/nuccore/80 … mask=65536
XP_002382247: https://www.ncbi.nlm.nih.gov/nuccore/238502026
OLP98873: https://www.ncbi.nlm.nih.gov/nuccore/1129194414
CDW76368: https://www.ncbi.nlm.nih.gov/nuccore/678335661
AHB99005: https://www.ncbi.nlm.nih.gov/nuccore/56 … mask=65536
OAQ27448: https://www.ncbi.nlm.nih.gov/nuccore/1032646064
OUM69730: https://www.ncbi.nlm.nih.gov/nuccore/1198313040
OGL53990: https://www.ncbi.nlm.nih.gov/nuccore/1084158629
EIE92391: https://www.ncbi.nlm.nih.gov/nuccore/76151980
XP_001590524: https://www.ncbi.nlm.nih.gov/nuccore/156049114
OTG33229: https://www.ncbi.nlm.nih.gov/nuccore/CM … =307832447
OLQ06943: https://www.ncbi.nlm.nih.gov/nuccore/1129204435
XP_001895277: https://www.ncbi.nlm.nih.gov/nuccore/170580473
XP_001584576: https://www.ncbi.nlm.nih.gov/nuccore/156030497
KOF84884: https://www.ncbi.nlm.nih.gov/nuccore/918315370 Возможно ли методом РСА определить
структуру этих участков белковых молекул? Если да, то нужен прайс. С уважением, Руководитель лаборатории Наномир, Александр Кушелев Второй вариант письма: Тема: Заказ
структуры белка Уважаемый ***** !
Прошу Вас рассмотреть нашу заявку на определение структуры белка: https://www.ncbi.nlm.nih.gov/nuccore/XM … port=fasta и выслать мне предложение и прайс-лист для заключения договора
с Вашей лабораторией. Связь со мной по e-mail С уважением, Обсуждение
Программная аллотропия
белков опровергает гипотезу фолдинга Материал с форума лаборатории Наномир: Кушелев: Уважаемая Виктория! Как Вы прокомментируете открытие программных спиралей белковых молекул?  Здесь Вы видите 19 вторичных структур поливалина. На схеме вторичной структуры представлены фундаментальные спирали: альфа-, 310-, пи-,
бета-
и программные:
14-, 35-, 352-, 144-, 25-, 22225-, 411412-, 255-, 2444-, 1144-, 11444444-, 23-, 335(коллагеновая)- Помните, как мы с Вами дискутировали по поводу лизиновых
"хвостов", т.е. участков пи-спиралей длиной более 10 аминокислотных остатков? Вы ещё писали, что это всё гипотетические структуры... А как Вы прокомментируете тему: "Рекорды
сверхдлинных спиралей белковых молекул"? Там встречаются фундаментальные спирали (альфа-, 310-, пи-, бета-) длиной более 3000 аминокислотных остатков. И программные спирали, в т.ч. фрактальные более 100 периодов, например, по 35 аминокислотных остатков в периоде, т.е. более 3500 аминокислотных остатков в
спирали. Если бы была верна гипотеза фолдинга, то зачем было бы кодировать все эти спиральные структуры? Антигомология =
программная аллотропия опровергает гипотезу фолдинга. Согласны?
Виктория Соколик: в конформации
белка закодированные спиральные участки не выстраиваются нескончаемой трубой друг за другом, а благодаря межмолекулярным взаимодействиям контактируют друг с другом, образуя энергетически равновесную, устойчивую конформацию с гидрофобным ядром. При этом, если отдельные закодированные спиральные участки в белке не получают дополнительной фиксации со стороны ближнего окружения атомов в радикалах аминокислот или остаются экспонированы в гидрофильную среду, то они как правило разворачиваются под действием
молекул воды и образуют уже не свернутые или петлевые области в структуре белка. Вышеописанный процесс носит название ФОЛДИНГ белка. Кушелев: Уважаемая и гениальная Виктория! Вы можете прокомментировать эти две структуры полилизина? https://www.ncbi.nlm.nih.gov/nuccore/XM … port=fasta 
https://www.ncbi.nlm.nih.gov/nuccore/700454480 
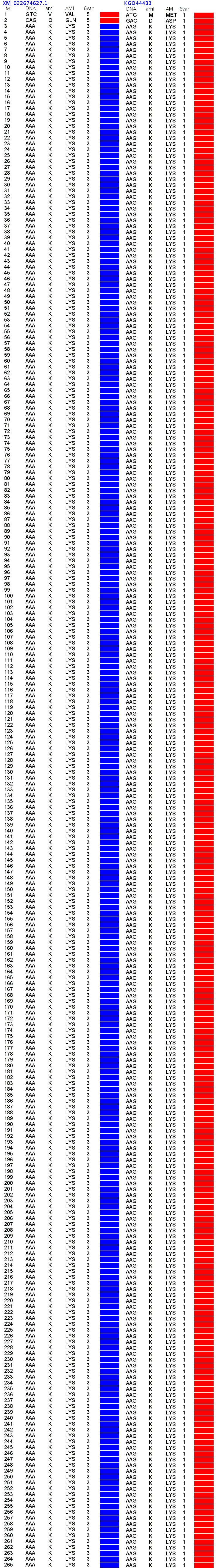
Кстати, какую структуру (или структурный шаблон, как Вы это называете) показывает Ваш алгоритм для этих двух белков? Подробнее Виктория Соколик: Выложите досье на один, самый интересный для Вас белок, и мы обсудим его структуру, декодированную Вами и в моей программе. Кушелев: Давайте начнём с первого: https://www.ncbi.nlm.nih.gov/nuccore/XM … port=fasta >XM_022674627.1 PREDICTED: Astyanax mexicanus protein FAM133-like (LOC111193520), partial mRNA
GTCCAGAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
Виктория Соколик: "программные" спирали являются комбинацией альфа-, 310-, пи-спиралей. Кушелев: Если Вы признаёте существование программных спиралей, то 1. Во-первых, почему они не изображаются у Вас на уровне 2D-структуры?
2. О каком фолдинге
может идти речь, если 3D модель программных спиралей строится по таблице композиционного кода? 
Виктория Соколик: Специфика 3/10, пи- и альфа-спиралей формируется уже в ходе процесса фолдинга на основе простейшего структурного шаблона 3/10
спирали. Кушелев: А тут подробнее, пожалуйста. Вот вторичная структура двух полилизинов: Вот, что показывает для пи-спирали алгоритм Кушелева и Соколик: 
Ваш алгоритм 2D ничего не показывает. Откуда при 3D моделировании у Вас появляется пи-спираль? Каков конкретный алгоритм Вашей 3D программы, который строит пи-спираль по коду: >XM_022674627.1 PREDICTED: Astyanax mexicanus protein FAM133-like
(LOC111193520), partial mRNA
GTCCAGAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA
AAAAAAAAAAAAAAAAAAAAAAAAA Почему один и тот же полилизин с кодом n(AAA) по Вашему 3D алгоритму фолдирует из 310-спирали в пи-спираль, а тот же полилизин с кодом n(AAG) из
310-спирали по Вашему 3D алгоритму фолдирует в прямую альфа-спираль. Что за чудо-алгоритм такой? Расскажите подробнее, как он работает для случая n(AAA) и n(AAG) ? В моём алгоритме всё предельно просто. По коду AAA из таблицы композиционного кода извлекается композиционный код пи-спирали и синий
"кирпичик". По коду AAG - композиционный код альфа-спирали и красный "кирпичик". Вы писали, что "мы получим одинаковый результат, но разными способами". Вот мне и любопытно, как конкретно по коду n(AAA) вы сначала получаете 310-спираль, а потом "волшебным фолдингом" из
310-спирали пи-спираль. Ну и при этом если код n(AAG), то "волшебный фолдинг" из той же 310-спирали-шаблона делает не пи-спираль, а альфа-спираль. Что это за волшебство, если не секрет, конечно... Обсуждение
Создан онлайн сервис "Определение структуры белка по нуклеотидной последовательности" Kushelev's PicoShop Проект для Бумстартера 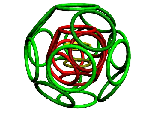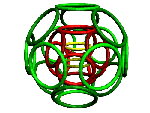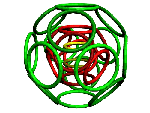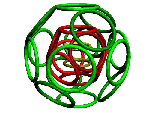   
На базе научного открытия нами создан онлайн-сервис по определению структуры белковых молекул.
Теперь мы сможем зарабатывать вместе. По старой технологии определение одной структуры белка обходится примерно в 10 000 евро, а ждать нужно от 2 месяцев до 3 лет. По новой технологии структура определяется в 1000 раз точнее и в миллион раз быстрее. 80% от найденного Вами заказа принадлежат Вам, как менеджеру. Наш лозунг: "В 1000 раз лучше и в 1000 раз дешевле!" Ваша задача заключается в размещении рекламы на онлайн-сервис белковых структур. Рынок этих структур очень
большой и продолжает стремительно расти. Ежедневно кто-то оплачивает до 60 структур по средней цене 10 000 евро за штуку. Новая технология позволяет на одном персональном компьютере обслужить до 100 заказчиков в день. При этом качество результата, полученного по новой технологии в 1000 раз выше по точности, в миллион раз по быстродействию и в 30 раз шире по номенклатуре белковых молекул. Единственное, что нам сегодня не хватает - рекламы. Как получить Вашу первую зарплату менеджера? Найти заказчика белковых структур и убедить его заказать за счёт лаборатории Наномир пробный заказ. Когда заказчик распробует новую технологию, он начнёт делать коммерческие заказы. С первого коммерческого заказа менеджер получает 80%. С последующих заказов процент будет постепенно уменьшаться, но с первого заказа другого заказчика менеджер снова получит 80%. Зарплата менеджера может достичь миллиона евро в день. И это не
предел.   
Первые 100
заказов за счёт лаборатории Наномир. Обсуждение
Инвестирование научных проектов
Виктория Соколик: Уважаемые коллеги, Вашему вниманию предоставляется услуга -- моделирование 2D и 3D структуры любого белка без ограничений в его размере и степени изученности с помощью программного обеспечения, базирующемся на принципиально новом подходе декодирования нуклеотидной последовательности, детерминирующей данный белок. Всё,
что необходимо от заказчика, это нуклеотидная последовательность мРНК интересующего его белка (или код этой нуклеотидной последовательности в EMBL, или хотя бы код самого белка в PDB). В течение 1-3 суток мы готовы предоставить Вам схему вторичной структуры заказанного белка (2D), модель его пространственной структуры (3D) в виртуальном пространстве,
а также файл .pdb с координатами каждого атома белка. Файл .pdb может быть использован по аналогии с файлами закристаллизованных белков из PDB банка для дальнейшего конформационного анализа белка методами молекулярной динамики с учётом физико-химической специфики микроокружения белка или его взаимодействия с лигандами. Таким образом, Вы сможете максимально быстро удобным для Вас способом (по электронной почте, на сайте либо на электронном носителе) получить информацию о структурном шаблоне Вашего белка. Сотрудничество может быть различным: - участие в научных дискуссиях на форуме (конструктивное) - совместное создание коммерческого продукта - поиск инвесторов - выступить менеджером по продаже
готовых коммерческих продуктов - конструктивные предложения по продвижению идей лаборатории Наномир - содействие в проведении экспериментов и т.п.
- написание совместных научных статей и т.п. - материальный
вклад (денежный или обеспечение оборудованием и материалами)
Пожалуйста, сообщайте о своем вкладе, чтобы мы зачли Вас как партнера лаборатории Наномир. +7-926-5101703, +7-903-2003424, +7-916-8265031, Skype: Kushelev2009, mail: kushelev20120@yandex.ru
веб-мани: WM-кошелек
R426964799301 Другие способы перевода Огромное спасибо всем за помощь и поддержку!
|